阻害剤KNI-272が結合したHIV-1プロテアーゼの中性子結晶構造解析
数日前に以下のニュースを読んで以来,PDBデータ2zyeおよびそのPDBsumデータと格闘してどうにかあれこれ画像を作成することができた。
阻害剤KNI-272が結合したHIV-1プロテアーゼの構造で,これ自体過去にも提出されていたが,上記プレスリリースにあるように中性子回折によるものなので水素(あるいは重水素)原子の位置も示され,厳密な位置関係が確認できる。ただ複数の座標が列記されていたほかJmolでは不要な結合が表示されたりで,試行錯誤で一部H・D原子を添削するというデータ加工をする必要があった。以下でご覧いただきたい。
- Jmolで見るトピックス分子(生活環境化学の部屋) | PDBsumのLigand-SITE情報 | HIVとエイズ(他のKNI-272結合したHIV-1プロテアーゼデータも掲載)
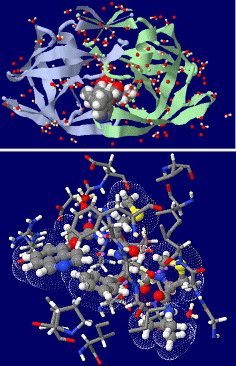
阻害剤KNI-272結合HIV-1プロテアーゼの中性子構造解析データ2zye
〔上〕全体構造,〔下〕一部H・D原子を追加したPDBsumデータ
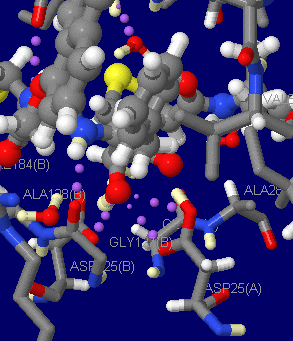
上掲プレスリリース図2の該当部分(Asp25とAsp125が触媒残基とされる)
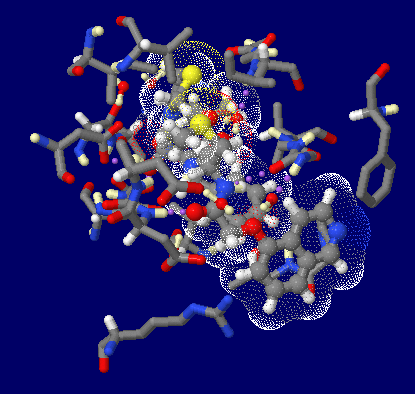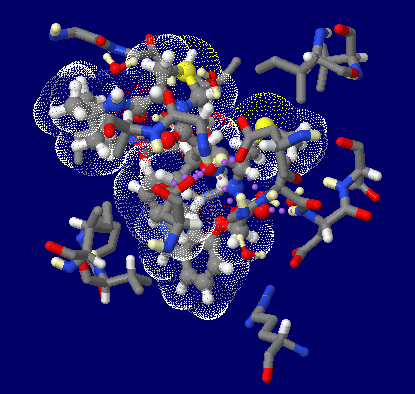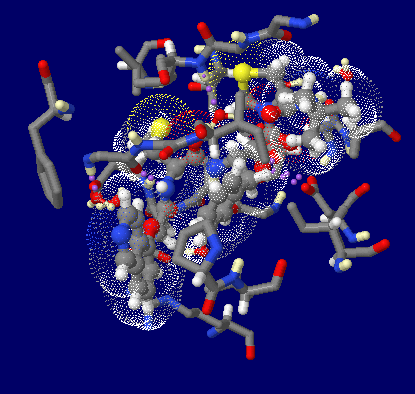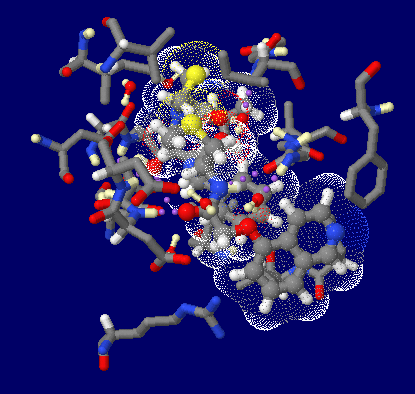
加工PDBsumデータのアニメ画像
鍵と鍵穴に介在する水分子の位置もわかり,複雑な生体分子内の詳細なありさまを観察できるのはとても興味深い。そこにおいて分子ビューアJmolの役割は大きいと思う。
なお,HIV-1プロテアーゼについては以下も参照されたい。
